Genomas
- DNA:
- 4 bases: A, G, C, T
- G liga com C, A liga com T
- Dupla hélice (complementares)
- RNA:
- 4 bases: A, G, C, U
- G liga com C, A liga com U
- Síntese de proteínas
Sequenciamento de genomas
- Sanger (1977)
- Leituras longas (400-700 bp)
- Usado para o sequenciamento do genoma humano (3 Mbp)
- Custo para 1M bp: US$ 2400
- Shotgun sequencing
- Quebrar cadeias longas de DNA
- Remontar a cadeia original computacionalmente
- Tipicamente cobertura de 6x
- Next generation sequencing
- leituras curtas (50-400 bp)
- alta cobertura (10-100x)
- Custo para 1M bp: US$ 0.05 a US$ 10
- Porém custo computacional dispara
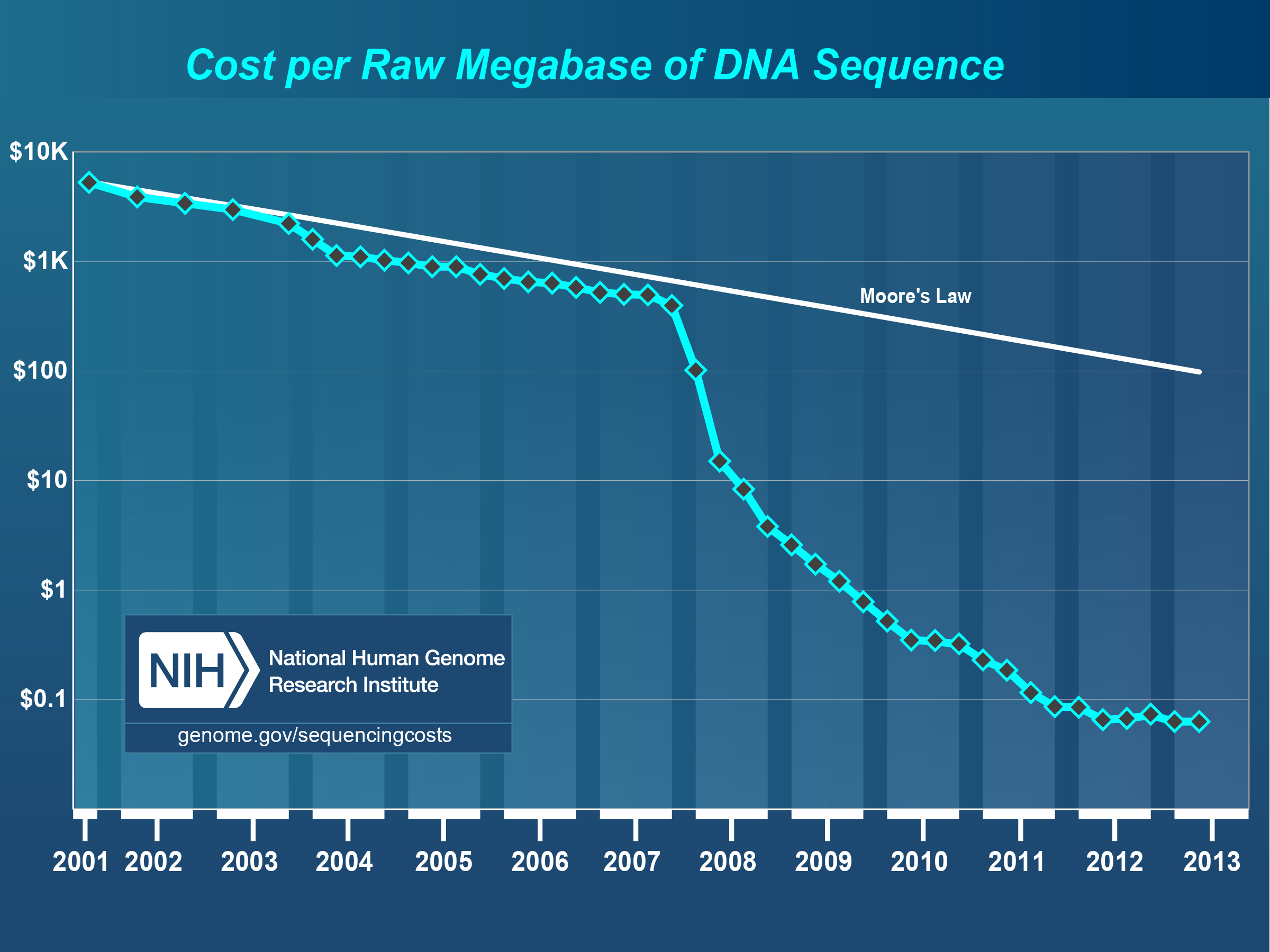
Custo por megabase
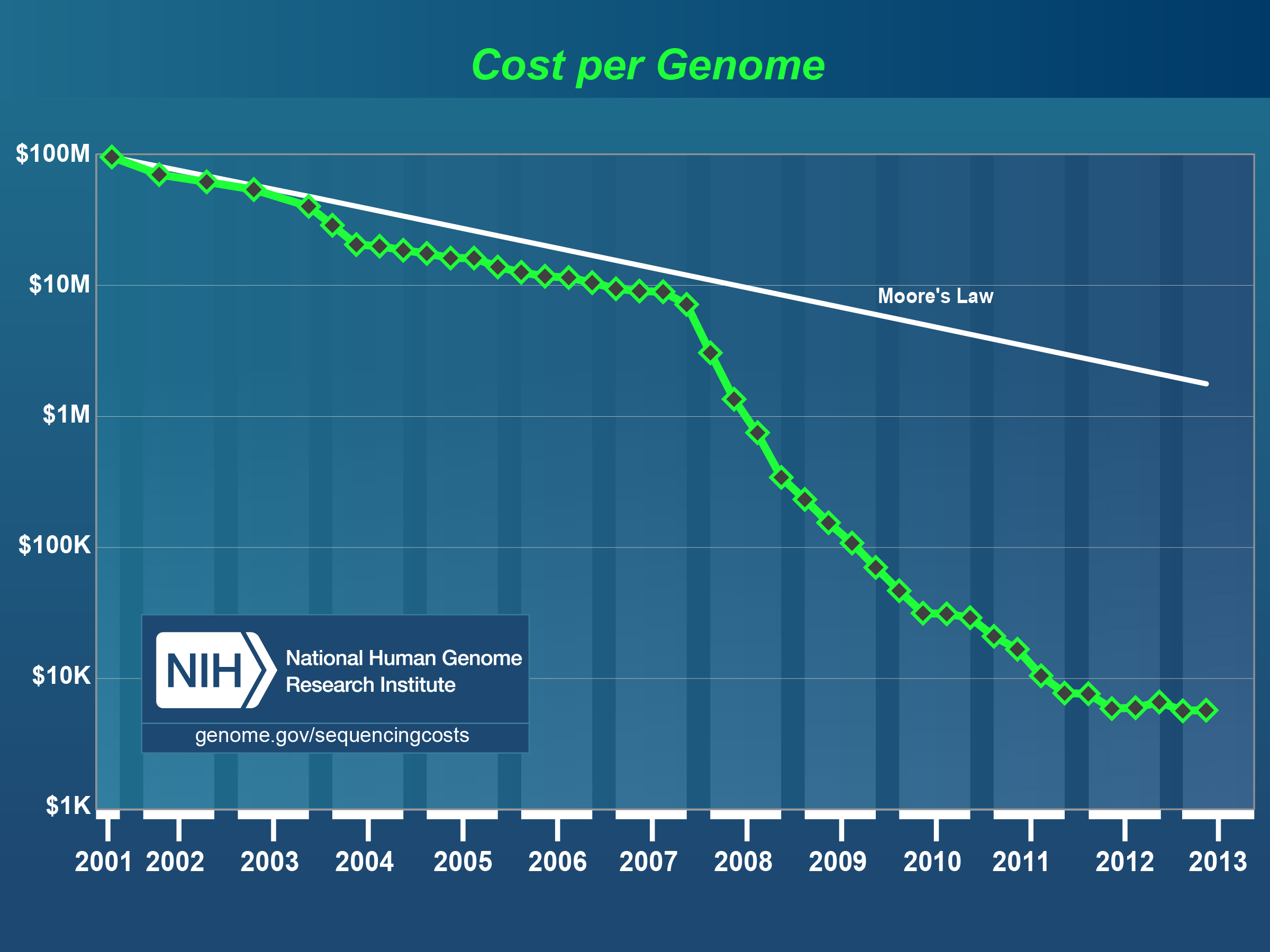
Custo por genoma
Bioinformática
- Computação aplicada a problemas biológicos
- Principalmente genômica e relacionados
- Uma via de duas mãos:
- Algoritmos clássicos de strings, grafos, programação dinâmica
- Problemas forçando a criação de novos algoritmos
- Principalmente devido à escala
Rosalind
- Plataforma para aprendizado através de solução de problemas
- Inspirado pelo Project Euler e Google Code Jam
- Nome em homenagem a Rosalind Franklin
- Cristalografia de raios X
Escolhendo problemas
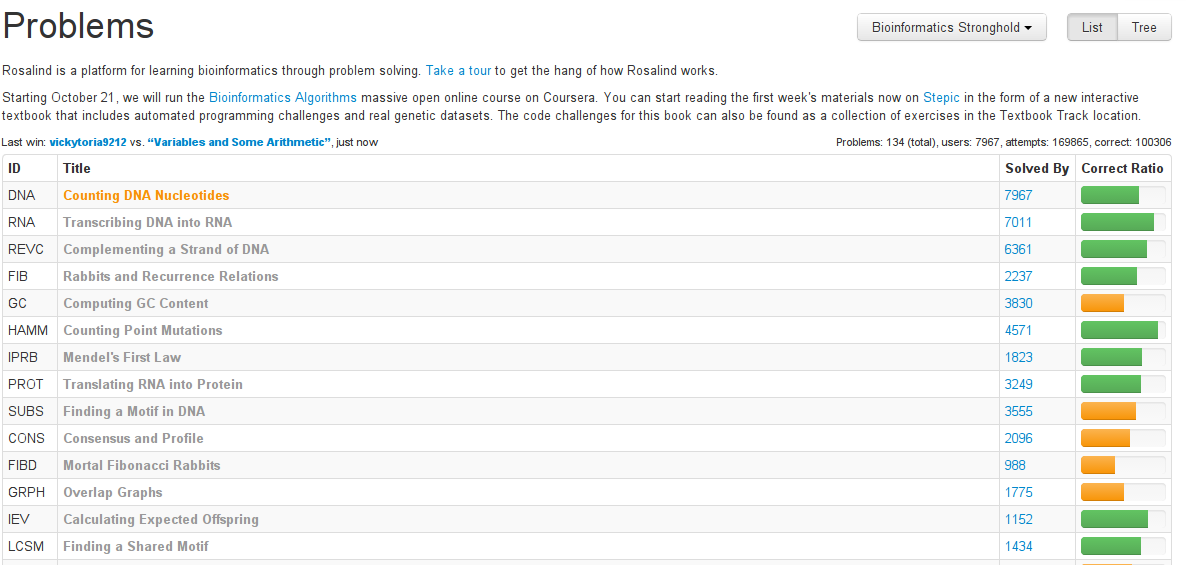
Lista
Escolhendo problemas
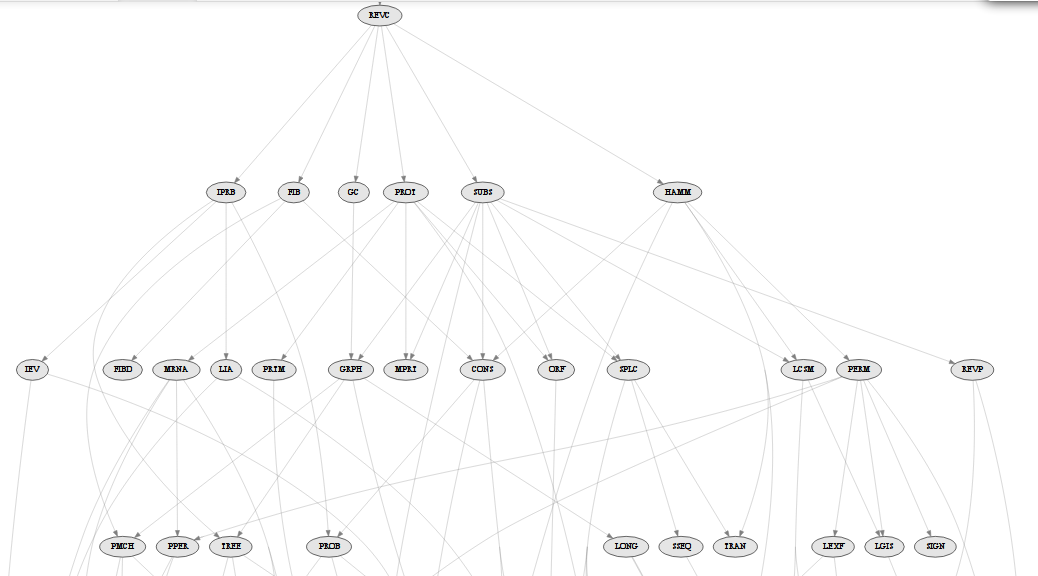
Árvore
Escolhendo problemas
Tópicos
Escolhendo problemas

Locais
Gamificação!
Anatomia de um problema
- http://rosalind.info/problems/dna/
- Descrição
- Especificação da entrada
- Especificação da saída
- Entrada exemplo
- Saída exemplo
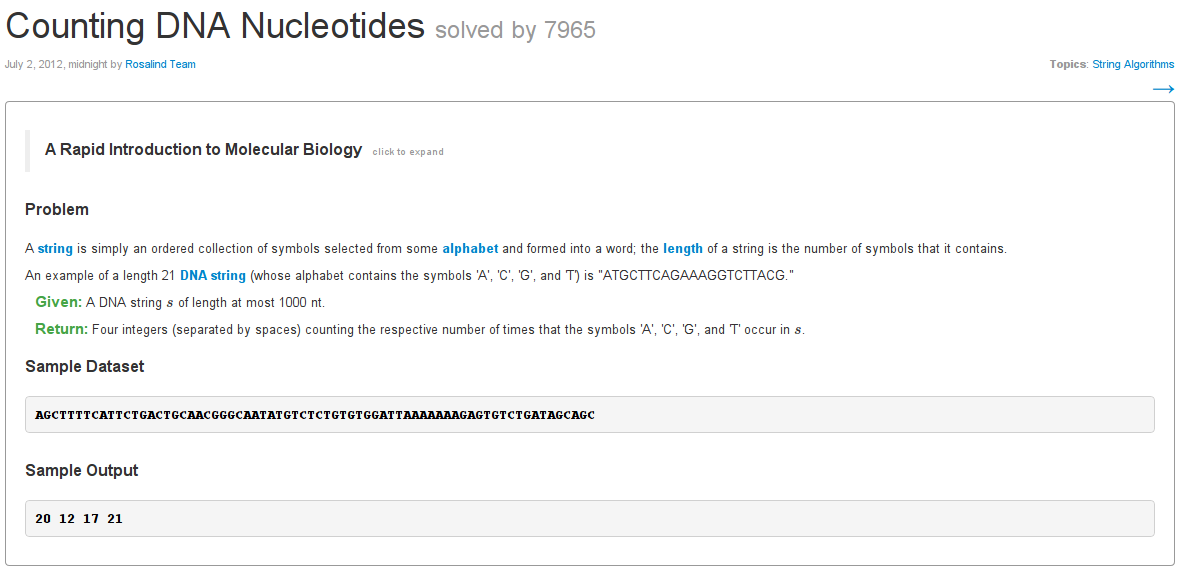
Anatomia de um problema
Submissão
Não ignore a explicação!
Aprendizado
- Python 2/3
from __future__ import print_function, division
- Cuidados com dict (items, values e keys retornam views no Python 3)
- from functools import reduce
- List comprehensions vs map/filter
List comprehensions provide an alternative to using the built-in map() and filter() functions. map(f, S) is equivalent to [f(x) for x in S] while filter(P, S) is equivalent to [x for x in S if P(x)]. One would think that list comprehensions have little to recommend themselves over the seemingly more compact map() and filter() notations. However, the picture changes if one looks at a more realistic example. Suppose we want to add 1 to the elements of a list, producing a new list. The list comprehension solution is [x+1 for x in S]. The solution using map() is map(lambda x: x+1, S). The part “lambda x: x+1” is Python’s notation for an anonymous function defined in-line.
It has been argued that the real problem here is that Python’s lambda notation is too verbose, and that a more concise notation for anonymous functions would make map() more attractive. Personally, I disagree—I find the list comprehension notation much easier to read than the functional notation, especially as the complexity of the expression to be mapped increases. In addition, the list comprehension executes much faster than the solution using map and lambda. This is because calling a lambda function creates a new stack frame while the expression in the list comprehension is evaluated without creating a new stack frame.
http://python-history.blogspot.com.br/2010/06/from-list-comprehensions-to-generator.html
- return map(dna.count, order)
+ return [dna.count(o) for o in order]-from itertools import starmap
-import operator
def hamming(s1, s2):
- return sum(starmap(operator.ne, zip(s1, s2)))
+ return sum(a != b for a, b in zip(s1, s2))- Usando context manager para lidar com arquivos
with open('rosalind_dna.txt') as dataset:
dna = dataset.readline().rstrip()
- Sets FTW!
with open(os.path.join('data', 'rosalind_dbru.txt')) as dataset:
dna_set = {s.rstrip() for s in dataset}
reverse_complements = {reverse_complement(s) for s in dna_set}
dna_strings = dna_set | reverse_complementsNovidades
- https://www.coursera.org/course/bioinformatics
- Começa dia 21
- Livro-texto disponível:
https://beta.stepic.org/Bioinformatics-Algorithms-2/
Obrigado
luizirber.org
luiz.irber@gmail.com